
Com a evolução da pandemia causada pela Covid-19, os testes diagnósticos também evoluíram consideravelmente, tanto para o diagnóstico de infecção ativa, como os testes moleculares de RT-PCR, quanto para a pesquisa de imunidade adquirida ou diagnóstico complementar através de anticorpos séricos.
Na parte de testes de RT-PCR, as novidades ficam por conta dos testes mais rápidos e também da maior capacidade de produção através de novas metodologias. Uma das novidades fica por conta do teste Xpert Xpress (Cepheid, EUA), realizado no equipamento GeneXpert, o mesmo que já era utilizado para outros testes moleculares rápidos como a pesquisa de M. tuberculosis, Influenza vírus e Clostridium difficile, dentre outros. Para quem ainda não está familiarizado com este tipo de teste, ele é todo realizado dentro de uma espécie de cartucho aonde ocorre todo o processo de amplificação por RT-PCR, através de um equipamento que coordena toda a parte mecânica do teste e análise dos resultados. Após concluído, o cartucho é descartado. O resultado é disponibilizado em até 24h da realização do exame. São utilizados 2 genes alvos, gene E e gene N. O gene E é comum ao SARS-Cov e o gene N é específico para o SARS-CoV-2.
A grande maioria dos estudos disponíveis sobre a performance, mostram que o grau de sensibilidade é semelhante ao dos testes convencionais (tab.1). As indicações incluem pacientes que precisam fazer procedimentos hospitalares de emergência em ambiente seguro, e pacientes de maior risco ou com quadro clínico que requer maior cuidado e precisa do diagnóstico com urgência.
Tabela 1. Relação de estudos comparativos entre Xpert Xpress SARS-CoV-2 e outras metodologias.
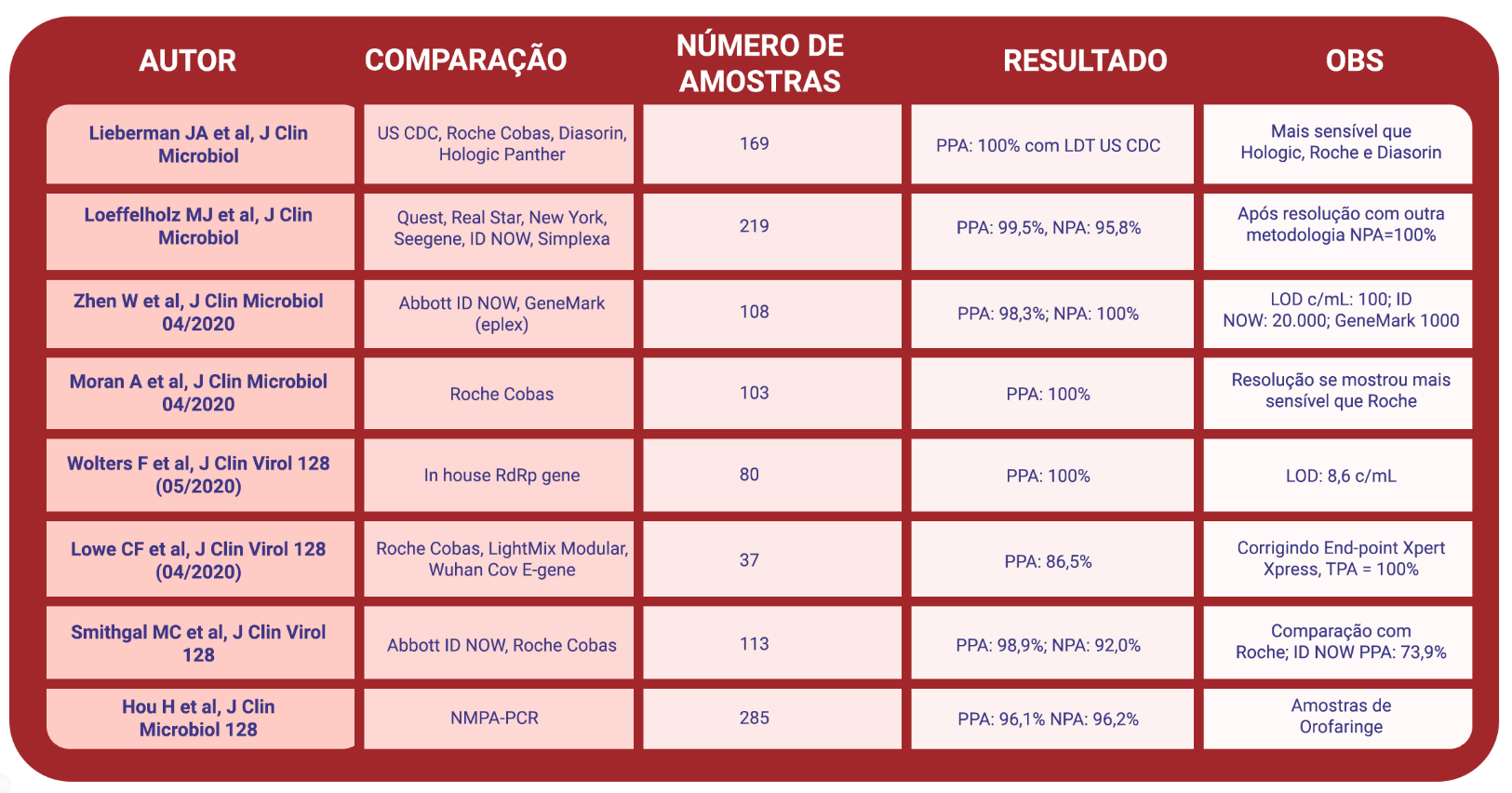
PPA: Positive Percentual Agreement; NPA: Negative Percentual Agreement; LOD: Limit Of Detection.
No processo de validação interna do teste, comparamos o GeneXpert SARS-CoV-2 com um teste de RT-PCR tradicional, utilizando plataforma automatizada, Allplex 2019-nCov, Seegene (Coreia do Sul), entre 10 amostras, sendo 5 positivas e 5 negativas, obtendo PPA e NPA de 100% para ambos.
Das 5 amostras positivas, 2 tinham CT alto, correspondendo a baixa carga viral (37,62 para o gene RdRp e 39,29 para gene N). Em seguida foi feito um pool de amostras positivas e feito diluições seriadas, cujos resultados foram concordantes até a diluição de 1:10.000), comprovando PPA de 100% e sensibilidade semelhante entre as duas técnicas, mesmo em amostras diluídas.
Em relação aos testes para pesquisa de anticorpos, houve também grande evolução, principalmente com a disponibilização de novos testes, principalmente em plataformas automatizadas, que permitem maior segurança e rapidez na entrega dos resultados. Os principais podem ser listados, como Abbott Arquitect IgG, EUA (N), Ortho Vitros IgG, EUA (S), Diasorin Liaison IgG, Itália (S1/S2) e Roche Cobas Total (N), Suíça; além dos que já vinham sendo utilizados como Euroimmun (Alemanha) IgG, IgA (S), IgG NCP (N) e Snibe Maglumi (China) IgG e IgM. O que existe de diferença nos testes, além da sensibilidade e especificidade analítica é o substrato utilizado como antígeno para a captura de anticorpos, mas, segundo a grande maioria dos estudos o maior grau de sensibilidade é obtivo após a 3a semana após o início da sintomatologia em todos os testes.
O vírus SARS-Cov-2 apresenta uma cadeia simples de RNA, que está estreitamente relacionado ao SARS-CoV e outros membros da linhagem B do genogrupo dos coronavírus, como o OC43, HKU1, 229E e NL63. O RNA do coronavírus codifica para quatro categorias principais de proteínas estruturais: Membrana (M), Envelope (E), Nucleocapsidio (N) e Spike (S). A proteína N, que induz à imunidade mediada pelos linfócitos CD8+, é amplamente utilizada como antígeno para a captura de anticorpos nos testes reagentes, por ser de fácil manipulação e apresentar bom grau de especificidade. Já a proteína S, está intrinsicamente ligada à ação viral, pois é uma proteína de fusão viral, que desempenha papel crucial na infecção, ao reconhecer os receptores da célula hospedeira. A entrada na célula é mediada pela proteína S transmembrana que forma homotrímeros que se projetam para fora da superfície viral. A proteína S inclui duas subunidades funcionais, S1 e S2, sendo a primeira responsável pela ligação ao receptor da célula hospedeira e a segunda pela fusão das membranas viral e celular.


As proteínas S e N do coronavírus são componentes imunogênicos importantes, sendo produzidas em quantidades abundantes durante a infeção. A proteína S é o principal determinante de imunidade protetora e de transmissão dos coronavírus; os anticorpos monoclonais contra a proteína S podem neutralizar a infectividade viral14. Em um estudo comparativo13 de anticorpos contra o antígeno S1/S2 do teste Diasorin Liaison, com o teste de neutralização viral, foram obtidos índice de concordância com resultados positivos (PPA) de 94,4% e índice de concordância com resultados negativos (NPA) de 97,8%, confirmando que os anticorpos detectados por este determinado teste, que utiliza as proteínas S1/S2, apresentam função neutralizante contra o SARS-CoV-2. Apesar de dados que mostram que os anticorpos voltados à proteína N também podem ser neutralizantes, este teste, Liaison Diasorin, foi o primeiro a ter o efeito neutralizante bem documento.
Após a validação dos novos testes sorológicos, modificamos o protocolo de realização dos testes para a pesquisa de anticorpos contra o SARS-CoV-2, passando a usar 3 metodologias em paralelo para a confirmação de resultados (Snibe, Roche e Diasorin) e consequentemente, detectando anticorpos tanto para a proteína N quanto para a proteína S, conforme recomendação para se obter maior grau de sensibilidade e de especificidade.
1. RT-PCR utilizando teste Allplex Seegene (Coreia do Sul), usando como alvo três regiões gênicas, E, RdRp e N. - Resultados em 2 dias úteis (incluindo sábado). - Raspado de Nasofaringe.
2. RT-PCR teste rápido Xpert Xpress GeneXpert Cepheid (EUA) - Resultados em até 24h da realização do exame.. Caso o pedido esteja com descrição do exame incompleta, será feito o PCR normal e não o Xpress, que o resultado será de acordo com o exame realizado.
3. Pesquisa de Anticorpos IgM e IgG para SARS-CoV-2 usando 3 metodologias (Cobas Roche, Liaison Diasorin e Snibe) - Resultado em 2 dias úteis.
4. Pesquisa de Anticorpos IgG Neutralizantes (Liaison Diasorin) para SARS-CoV-2 - Resultado em 2 dias úteis.
Referências bibliográficas:
1. Wan, W, Xu, Y, Gao R, Lu R, Han K, Wu G, Tan W, Research Letter, JAMA on line, 11 March 2020 https://jamanetwork.com/journals/jama/fullarticle/2762997
2. Xu k et al, Factors associated with prolongated RNA COVID-19, Factors associated with prolonged viral RNA shedding in patients with COVID-19, Academic Ouput.com, 2020
3. Long Q-X et al, Antibody responses to SARS-CoV-2 in patients with COVID-19, Nature Medicine Brief Report, 2020 https://doi.org/10.1093/cid/ciaa351
4. Montesinosa I, Grusonb D, Kabambac B, Dahmaa H, Van den Wijngaerta S, Rezac S, Carboneb V, Vandenbergd O, Gulbise B, Wolfff F, Rodriguez-Villalobosc H, Evaluation of two automated and three rapid lateral flow immunoassays for the detection of anti-SARS-CoV-2 antibodies, J Clin Virol, 128(2020) 104413. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7198434/
5. Lou B, Li T-D, Zheng S-F, et al. Serology characteristics of SARSCoV-2 infection since exposure and post symptom onset. Eur Respir J 2020; in press (https://doi.org/10.1183/13993003.00763-2020).
6. Kortha J, Wildea B, Dolffb S, Anastasiouc OE, Krawczykb A, Jahna M, Cordesd S, Rosse B, Esserf S, Lindemanng M, Kribbena A, Dittmerc U, Witzkeb O, Herrmannc A, SARS-CoV-2-specific antibody
detection in healthcare workers in Germany with direct contact to COVID-19 patients, J Clin Virol 128(2020) 104437 https://doi.org/10.1016/j.jcv.2020.104437
7. Bao L, Deng W, Gao H, Xiao C, Liu J, Xue J, Qi L, Liu J, Yu P, Xu Y, Qi F, Li F, Xiang Z, Yu H, Gong S, Liu M, Wang G, Wang S, Song Z, Liu Y, Zhao W, Han Y, Zhao L, Liu W, Wei1 Q, Qin C, Lack of Reinfection in Rhesus Macaques Infected with SARS-CoV-2, BioRexiv 2020 https://doi.org/10.1101/2020.03.13.990226
8. Bullard J, Dust K, Funk D, Strong JE, Alexander D, Garnett L, Boodman C, Bello A, Hedley A, Schiffman Z, Predicting infectious SARS-CoV-2 from diagnostic samples, Clin Inf Dis, 22 May 2020, https://doi.org/10.1093/cid/ciaa638
9. Dias VMCH, Carneiro M, Vidal CFL, Corradi MFDB, Brandão D, Cunha CA, Chebabo A, Oliveira PRD, Michelin L, Rocha JLL, Waib LF, Carrilho CM, Lobo SMA, Oliveira MC, Nunes RR, Diego LAS, Santos AS, Muglia V, Souza Jr AS, Escuissato D, Neto CA, Chatkin JM, Martins R, Maurici R, Costa SF, Alves JS, Nascimento MM, Moura-Neto JA, Orientações sobre Diagnóstico, Tratamento e Isolamento de Pacientes com COVID-19, J. Infect. Control, 2020 Abr-Jun;9(2):XX-XX [ISSN 2316-5324], http://jicabih.com.br/index.php/jic/article/view/295
10. Lieberman JA, Pepper G, Naccache SM, Huang M-L, Jerome KR, Greninger AL, Comparison of Commercially Available and Laboratory Developed Assays for in vitro Detection 2 of SARS-CoV-2 in Clinical Laboratories, J. Clin. Microbiol. doi:10.1128/JCM.00821-20, https://jcm.asm.org/content/early/2020/04/27/JCM.00821-20
11. Loeffelholz MJ, Alland D, Butler –Wu SM, Pandey U, Perno CF, Nava A, Carroll, KC, Mostafa H, Davies E, et al, Multicenter Evaluation of the Cepheid Xpert Xpress SARS -CoV -2 Test, J. Clin. Microbiol. doi:10.1128/JCM.0092620, https://jcm.asm.org/content/jcm/early/2020/04/30/JCM.00926-20.full.pdf
12. Lowea CF, Matica N, Ritchiea G, Lawsona T, Stefanovica A, Champagnea S, Leunga V, Romneya MG, Detection of low levels of SARS-CoV-2 RNA from nasopharyngeal swabs using three comercial molecular assays, J Clin Virol 128 (2020) 104387, https://doi.org/10.1016/j.jcv.2020.104387
13. Bonelli F, Sarasini A, Zierolda C, Calleria M, Bonettib A, Vismarad C, Blockia F, Pallavicinia L, Chinali A, Campisid D, Percivalleb E, DiNapolib AP, Pernod CF, Baldantib F, Clinical And Analytical Performance Of An Automated Serological Test That 2 Identifies S1/S2 Neutralizing IgG In Covid-19 Patients Semiquantitatively, bioRxiv preprint doi: https://doi.org/10.1101/2020.05.19.105445
14. Liaison SARS-CoV-2 S1/S2 IgG package insert revisão 04 – 2020-0
